
导读
微生物组研究前沿从对微生物的形态描述转向揭示微生物群落共生的复杂机制。建立合成菌群(SynCom)是揭示群落装配过程的关键策略。然而,现有的方法主要是为了区分特定合成菌群中的特定微生物群体。为了解决这一问题,2024年发表于《Microbiome》的研究论文开发了差异荧光标记(DFM)的策略,通过流式细胞仪,成功地量化并追踪了SynCom在复杂条件下,在豌豆和大麦根际之间的定植组装动态。
主要研究结果
1. 构建DFM载体
为满足荧光显微镜和流式细胞仪检测所需蛋白量,同时避免由于过表达而导致的毒性,本研究选用pTn7-SCOUT作为质粒,将荧光蛋白mCherry、sYFP2和TagBFP构建到质粒,将重构的质粒转化到豌豆根瘤菌3841(Rlv3841)中,生成不同标记的菌株Rlv3841DFM菌株(Rlv3841R、Rlv3841Y、Rlv3841B、Rlv3841RY、Rlv3841RB和Rlv3841YB)
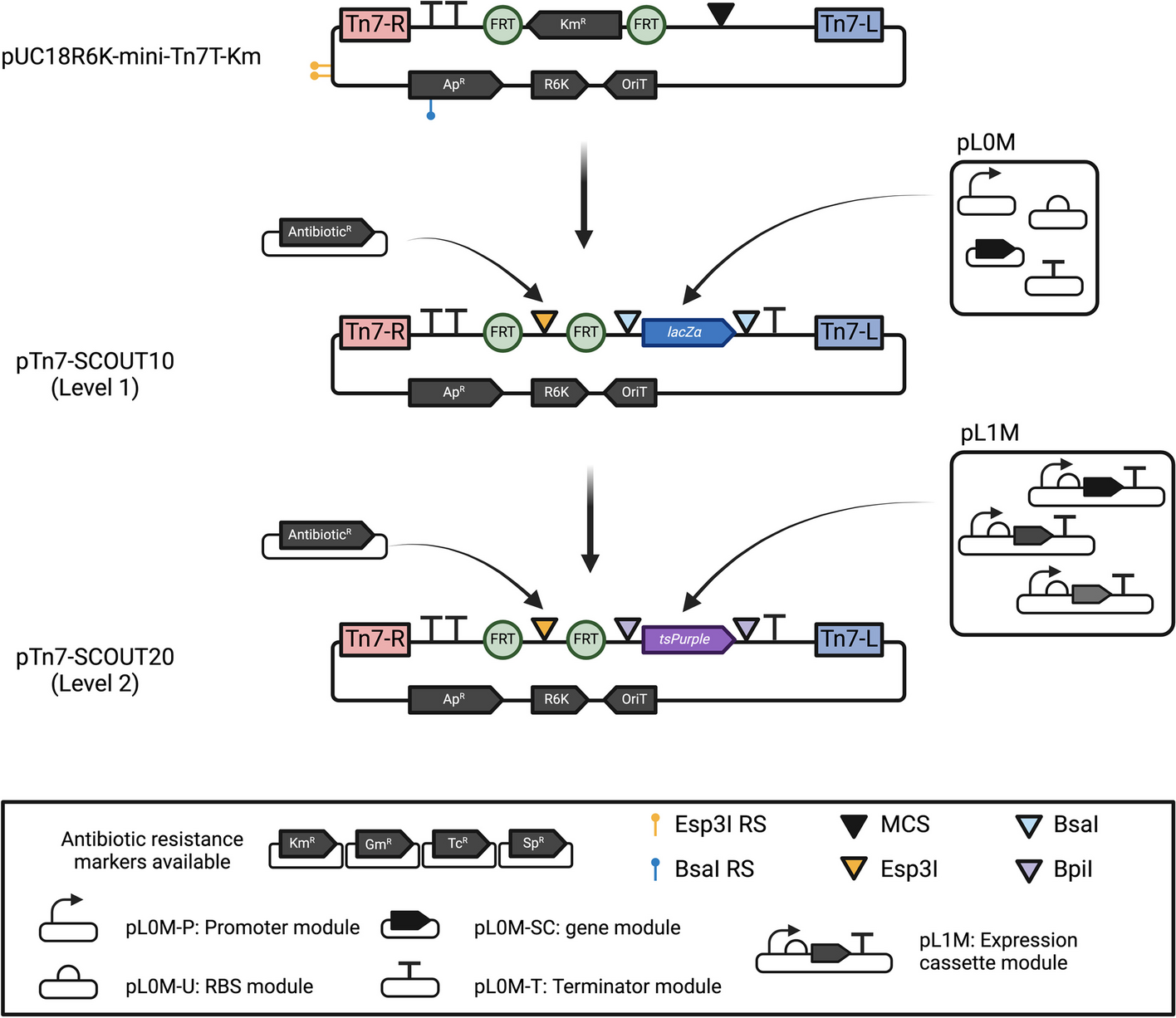
图1 pTn7-SCOUT载体构建
表1 用不同DFM组合标记的菌株描述
| Plasmid | AntbR | Description |
|---|---|---|
| pTn7-SCOUT10 | Ap | pL0V-Lv1-pUC18R6KT-miniTn7 with Esp3I sites for Antb modules |
| pTn7-SCOUT20 | Ap | pL0V-Lv2-pUC18R6KT-miniTn7 with Esp3I sites for Antb modules |
| pTn7-SCOUT11 | Ap-Gm | pL1V-Lv1-pUC18R6KT-miniTn7-Gm with BsaI sites for cloning |
| pTn7-SCOUT12 | Ap-Km | pL1V-Lv1-pUC18R6KT-miniTn7-Km with BsaI sites for cloning |
| pTn7-SCOUT13 | Ap-Tc | pL1V-Lv1-pUC18R6KT-miniTn7-TetAR with BsaI sites for cloning |
| pTn7-SCOUT14 | Ap-Sp | pL1V-Lv1-pUC18R6KT-miniTn7-Sp with BsaI sites for cloning |
| pTn7-SCOUT21 | Ap-Gm | pL2V-Lv2-pUC18R6KT-miniTn7-Gm with BpiI sites for cloning |
| pTn7-SCOUT22 | Ap-Km | pL2V-Lv2-pUC18R6KT-miniTn7-Km with BpiI sites for cloning |
| pTn7-SCOUT23 | Ap-Tc | pL2V-Lv2-pUC18R6KT-miniTn7-TetAR with BpiI sites for cloning |
| pTn7-SCOUT24 | Ap-Sp | pL2V-Lv2-pUC18R6KT-miniTn7-Sp with BpiI sites for cloning |
| pFlp-Km | Km | flp-cl-sacB-pL1V-Lv1-Km-pBBR1-ELT3 |
| pFlp-Gm | Gm | flp-cl-sacB-pL1V-Lv1-Gm-pBBR1-ELT3 |
| pFlp-Tc | Tc | flp-cl-sacB-pL1V-Lv1-TetAR-pBBR1-ELT3 |
2. DFM信号的安全性验证
通过计算世代时间确认DFM信号对豌豆根瘤菌3841没有危害。通过测试每种Rlv3841DFM菌株在豌豆根部单独接种和与Rlv3841U(未标记的菌株)竞争生长的能力,发现没有观察到显著差异,确认DFM不影响菌株竞争定殖的能力。证实了DFM结合流式细胞技术可以用于区分和定量样品中的六种细菌菌株,且对细菌的适应性没有影响。
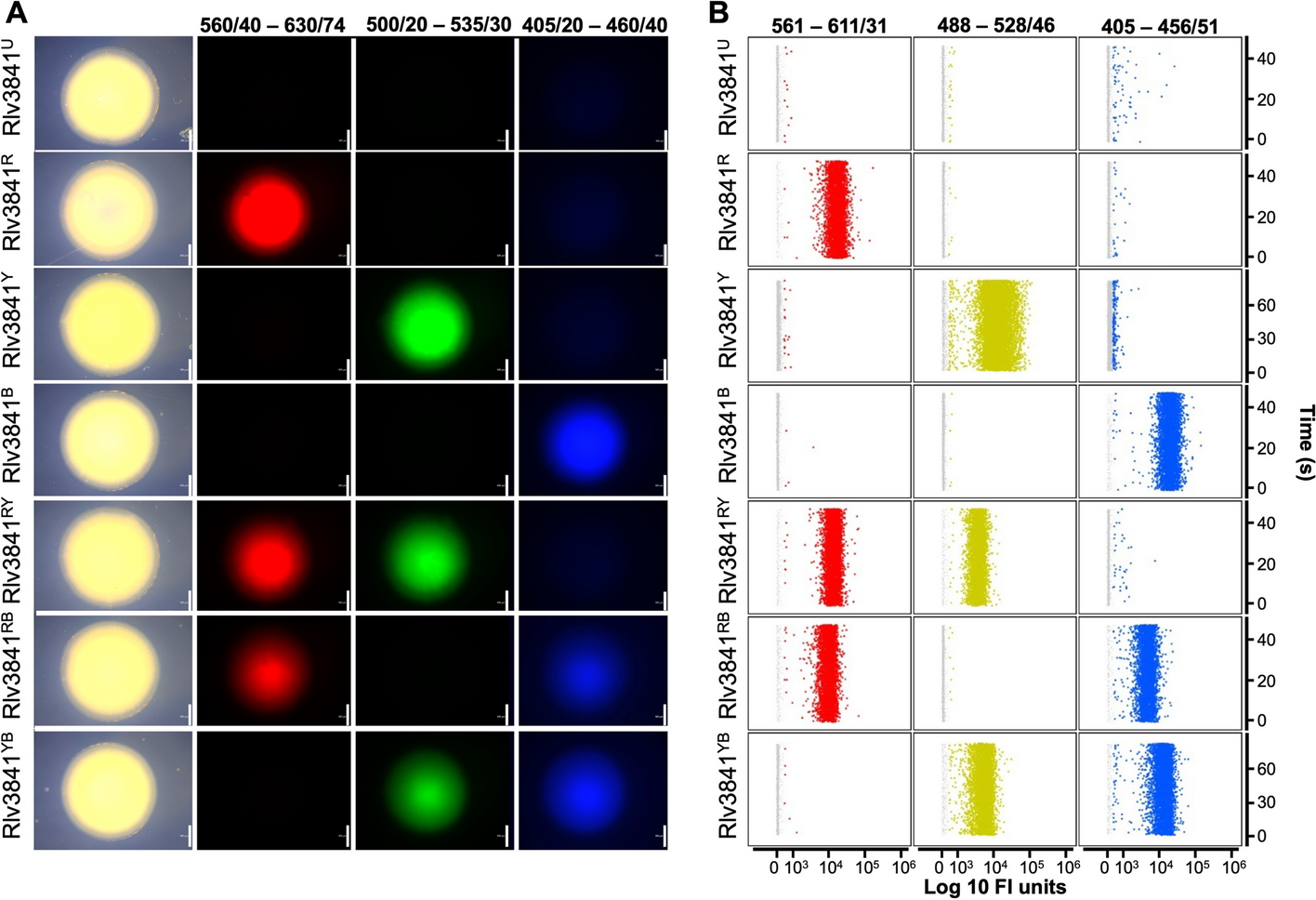 图2 细菌的差分荧光标记
图2 细菌的差分荧光标记
3. 利用DFM追踪合成群落中的细菌
本研究组装了一个模型SynCom(OxCom6),OxCom6群落的6个成员分别被DFM标记成特定的荧光(O.pituitosum AA2使用mCherry标记(OpAA2R),R.leguminosarum bv viciae 3841使用mCherry和mTagBFP标记(Rlv3841RB),A.olearius DQS-4使用sYFP2标记(AoDQS4Y),A.xylosoxidans AT1使用sYFP2和mTagBFP标记(AxAT1YB),E.cloacae AA4使用mCherry和sYFP2标记(EcAA4RY),P.luorescens SBW25使用mTagBFP标记(PfSBW25B)。分别在丰富营养培养基中、豌豆和大麦根中监测OxCom6的组装过程(监测时间分别为:4天、14天、14天),分析各个菌的实时丰度变化,实现每个OxCom6成员的绝对定量。
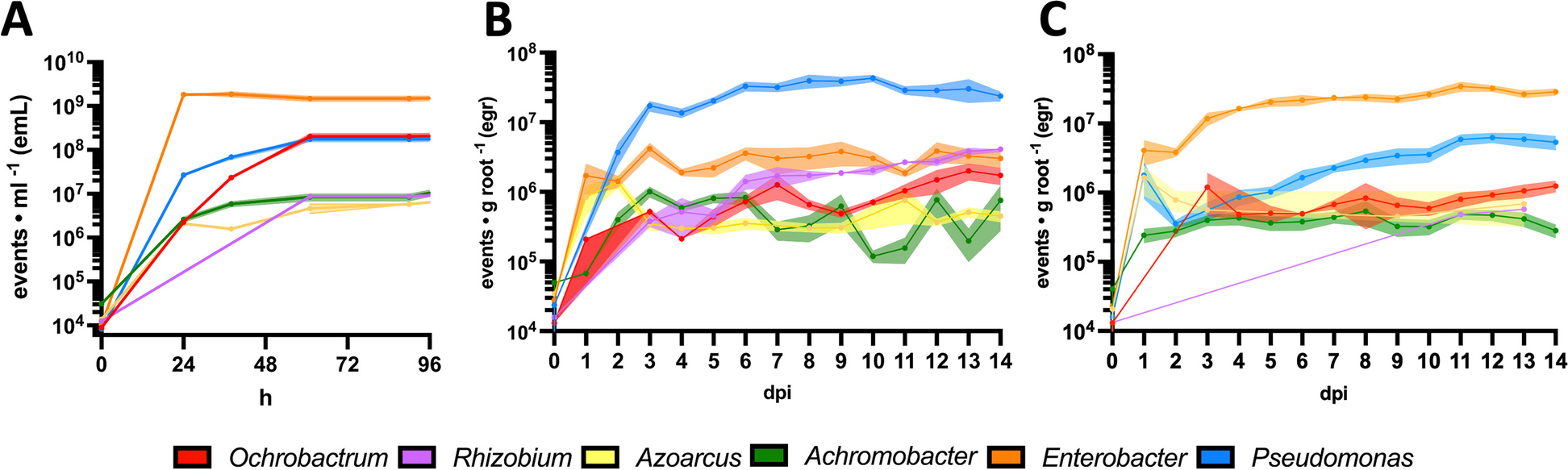 图3 利用DFM跟踪细菌合成群落
图3 利用DFM跟踪细菌合成群落
总结
本研究利用mCherry、GFP、YFP荧光信号对OxCom6中的6个菌株进行实时监测和定量,为添加到复杂样本中的菌株提供标记,为揭示微生物群落共生的机制提供重要思路,有望进一步探索并研究更大规模的微生物菌群动态。
本研究创新性地采用了荧光靶向DFM技术,作为追踪目标微生物的有力工具,为快速评估植物生态系统、动物体内环境及自然环境中微生物群落的组成与变化开辟了新的研究路径。然而,值得注意的是,为确保研究的准确性和有效性,任何被添加到实际环境或生物体(如肠道)中的菌株,都需经过有效的荧光标记处理,否则将难以实现对这些菌株的精确追踪与评估。
原文
https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-024-01792-2