一、什么是进化树
系统发育进化树 (Phylogenetic tree): 一般也叫系统进化树,进化树。它可以利用树状分支图形来表示各物种或基因间的亲缘关系。
分支系统发育分析 (Molecular phylogenetic analysis): 是用来研究物种或序列进化和系统分类的一种方法。一般研究对象是碱基序列或氨基酸序列,通过数理统计算法来计算生物间进化关系。最后,根据计算结果,可视化为系统进化树。
二、进化树中的构成
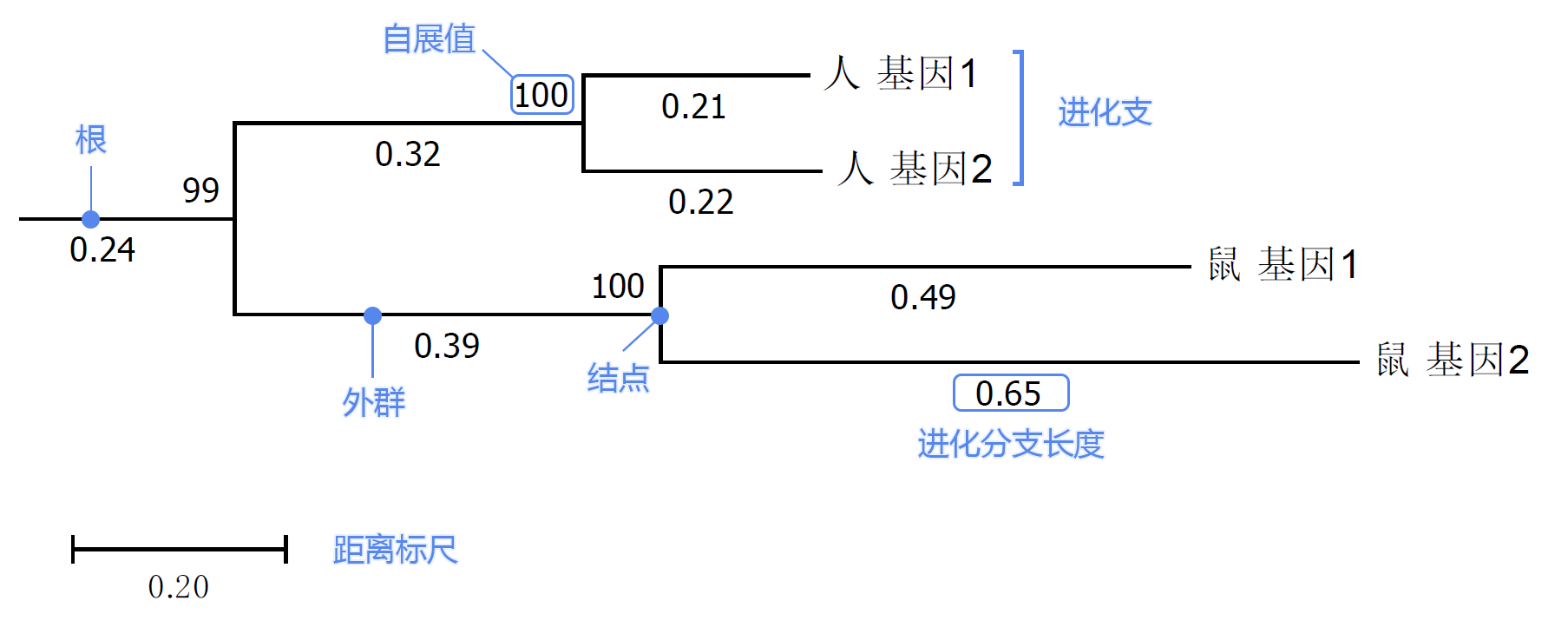
-
根 (Root)
所有分支的共同祖先
有根树-无根树
-
结点(Node)(注意与==节点==的区分,节点表示实体或事物。结点指交汇点)
每个结点代表一个分类单元,物种上可以是属,种群等,基因上可以是基因家族,同源物等。
另外一种解释:
node分为 外部节点与内部节点:外部节点又叫叶节点,也就是最外层的人基因1,人基因2等,代表参与分析的序列样本内部节点,也就是我们使用蓝色标注的位置,代表假定祖先。 -
进化支 (Branch)
也叫分支,指两种及以上的生物或序列组成的进化关系。
可以利用这个来看同源。比如,上图中人基因1与人基因2可能是旁系同源基因,而人基因1与鼠基因1可能是直系同源基因。
-
外群
与分析序列相关的生物序列,但是具有较远的亲缘关系。
-
进化分支长度
也叫遗传变异度,进化距离。一般会标注在分支线上,代表进化支变化的程度,越短代表差异越小,进化距离越近。比如人基因1与人基因2的遗传变异度为 0.21+0.22=0.43。
遗传变异度实际代表基因组序列中每个位点碱基的替换频率,计算方法也很简单:变异度=变异碱基数/总碱基数(%)。我们常见的形式,通常以0-1的小数来表示,代表100个碱基位点的变异度大小。
我们可以从水平方向上的分支及长度,看到进化谱系随着时间的变化,进化分支长度越长代表着该分支对应的物种或基因的变化越大。比如,对应上图,我们可以描述为人基因1相对其他基因在进化时间上更早,而且在进化时间上鼠基因2最晚。
有意思的是,根据基因序列相似度与进化时间假说对这种进化距离进行转换,就可以得到分子钟。比如,用它分析病毒进化树,甚至可以推断出初代病毒产生的时间点。
-
距离标尺
生物或序列间差异数值的单位长度,相当于进化树的比例尺。
-
Bootstrap value
一般会标注在结点,用来评估该分支的可信度。 Bootstrap value 对于我们后续分析比较重要,尤其在进化树评估中。
三、进化树评估
-
Bootstrap检验
对于进化树评估一般会使用 Bootstrap 进行检验。
Bootstrap检验,自举法检验,也叫自展,自助法。其实就是放回式抽样统计法的一种,通过对数据集多次重复取样,构建多个进化树,用来检查给定树的分枝可信度。
-
重复取样值
那么重复取样的b次数会在建树时设置,现在一般文章要求Bootstrap 取样值 >1000。
-
Bootstrap value 阈值
虽然根据严格的统计学概念,自展值需要要大于95%才较为可信。
然而在实际应用中,我们一般认为结点的 Bootstrap value > 70,这个分支就是可靠的。特别是==微生物等相似度比较大的分类中,一般大于50%就认为可信(小于50%不会显示)==。
-
Bootstrap value 与分支
如果低 Bootstrap value 更靠近分支末端,代表相似度太高而很难区分。
如果低 Bootstrap value 更靠近根,代表相似度太低。
四、几种进化树图
-
经典树图(Traditional)
可以清晰的展示出样本间进化距离和进化分支,但展示出来效果不炫。
-
圈图(Circle Tree)
本质是将树图极坐标化,在分析样本数量大的时候效果更佳,缺点是很难横向比对进化距离。适用于展示
差异较大的物种或基因样本。 -
辐射树(Radiation Tree)
这种图用于根不确定的进化树构建。它可以将相似度高的样本序列聚集在一起。因此,更适合做
亲缘关系近的物种或差异小的基因样本。
绘制进化树的工具有:ggtree、iTOL、GraPhlAn
参考资料:
进一步阅读: